CfMV mutaģenēze un vektoru konstruēšna
Lai iegūtu infekciozu CfMV cDNS vektoru, bija nepieciešams ievadīt cDNS 5 galā piemērotu saitu, kas ļautu maksimāli tuvu tam subklonēt 35S promotora sekvenci (skat. 22. lpp.).
CfMV infekciozās cDNS vektora veidošanai tika izmantotas divas plazmīdas, par vektoru izvēloties pJIT-60 (skat. att. 5). pJIT-60 satur divas 35S promotora sekvences, poli-A terminatoru un Amp rezistences gēnu. Kā vīrusa cDNS avotu izmantojām plazmīdas pUC57 atvasinājumu pXL 3-4, kas satur pilno cDNS kopiju zem T7 promotora (skat. att.6).
pXL 3-4 par izejas cDNS konstrukciju tika ņemta tāpēc, ka tā jau ir sevi pierādījusi kā potenciāla himēro vakcīnu kandidāte. No tās tika iegūta infekciju izraisoša RNS (A.Zeltiņš, nepublicēts), bet šāda metode ir tehniski sarežģīta un dārga. Tādēļ tiek meklēta iespēja iegūt infekciozu cDNS, kas būtu efektīvāka metode, lai iegūtu labāku rezultātu un arī lai ieguves process būtu ātrāks un ekonomiski izdevīgāks.
Lai ar PCR mutaģenēzes palīdzību CfMV cDNS 5 galā ievadītu KspAI un EheI saitus, ar attiecīgu praimeru (skat. 17. lpp.) palīdzību tika iegūts 0,6 kb fragments. Fragments tika sašķelts ar EheI un BamHI saitu un klonēts atpakaļ vektorā pXL 3-4, tādejādi iegūstot KspAI saitu cDNS 5' galā. Šo saitu var izmantot gan restrikcijas analīzei, gan kā saitu tālāku konstrukciju veidošanai.
Tā kā izejas plazmīda pXL 3-4 satur divus BamHI saitus, tad vispirms tika izveidota plazmīda p3-4K5', PCR produtu saligējot pa saitiem EheI un BamHI (skat. att. 7).
Miniprepu lizāti tika pārbaudīti, vai tie satur plazmīdas. Iegūtās plazmīdas apskatot ar elektroforēzes palīdzību uz 0,8 % agarozes gēla varēja redzēt, ka gandrīz visas plazmīdas ir neliela izmēra (skat. att. 8). Visas plazmīdas tika pārbaudītas ar divām restriktāzēm BamHI un KspAI, lai pārliecinātos vai ir nepieciešamais saits un vai ir īstais plazmīdas garums. No septiņiem paraugiem īstais bija paraugs Nr.1. (skat. att. 9).
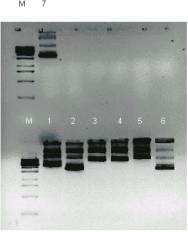
8.attēls. Miniprepu apskate 0,8 % agarozes 9.attēls. Paraugu pārbaude ar KspAI/BamHI, vai
gēlā uz plazmīdu p3-4K5 (M marķieris, dotie paraugi satur KspAI saitu (M marķieris, 1, 2,
1, 2, 3, 4, 5, 6, 7 paraugi) 3, 4, 5, 6, 7 paraugi, 1 pareizais klons)
Figure 8. Miniprep examination in 0,8% Figure 9. Examination of example with KspAI/
agarose gel for plasmid detection BamHI for KspAI site detestation (M marker, 1, 2,
p3-4K5 (M marker, 1, 2, 3, 4, 5, 6, 7 3, 4, 5, 6, 7 sample, 1 right clone)
samples)
Iegūtā plazmīda p3-4K5 un izejas plazmīda pXL 3-4 tika šķeltas pa saitiem BamHI un SalI (fragmentu garums attiecīgi ~3,0 kb un 3,5 kb) Abi fragmenti tika saligēti un iegūta plazmīda pXL 3-4K (skat. att. 10).
Miniprepu lizāti tika pārbaudīti, vai tie satur plazmīdu. Analizējot plazmīdas uz 0,8 % agaroze gēla varēja redzēt, ka tās ir dažāda lieluma. Tālākai analīzei tika paņemti 13 miniprepi, kas saturēja garākās plazmīdas (2., 4., 5., 10., 14., 15., 17., 19., 20., 21., 24., 25.). Tās tika pārbaudītas ar restriktāzēm ScaI, KspAI un BamHI (lai pārbaudītu vai plazmīda satur vajadzīgo saitu - KspAI). Šķeļot ar ScaI pareiza restrikcijas aina bija četrām plazīdām (14., 15., 17., 24.) (skat. att. 11).
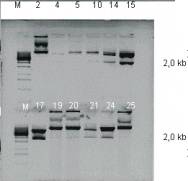
11.attēls. Miniprepu pārbaude uz orientāciju ar restiktāzi ScaI plazmīdai pXL 3-4K (M marķieris, 2, 4, 5, 10, 14, 15, 17, 19, 20, 21, 24, 25 paraugi, 14, 15, 17, 24 pareizās orientācijas pazmīdas)
Figure 11. Miniprep examination to orientation with restriktaze ScaI for plasmid pXL 3-4K (M marker, 2, 4, 5, 10, 14, 15, 17, 19, 20, 21, 24, 25 samples, 14, 15, 17, 24 right orientation plasmide)
Šīs četras plazmīdas tālāk tika grieztas ar KspAI un BamHI, lai pārliecinātos par KspAI saita esamību. Restrikcija uzrādīja to, ka visas četras plazmīdas satur KspAI saitu, par to liecināja pareizā garuma un daudzuma restrikcijas fragmenti (skat. att. 12).
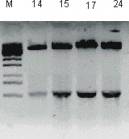
12.attēls. Pārbaude ar restiktāzēm KspAI/BamHI, lai pārliecinātos vai plazmīdas satur KspAI saitu (M marķieris, 14, 15, 17, 24 plazmīdas ar KspAI saitu)
Figure 12. Examination with restriktases KspAI/BamHI for detection of KspAI site in plasmids (M marker, 14, 15, 17, 24 plasmide with KspAI sit)
Ar sekvenēšanas analīzes palīdzību tika pārbaudītas ar PCR mutaģeēzes palīdzību vektorā ievadītais cDNS 5' reģions, kā ar tiešo (2.296), tā arī ar reverso (1.10) praimeri. Sekvenēšanai tika ņemtas plazmīdas pXL 3-4K un p3-4K5. Iegūtā sekvence atbilda datu bāzē atrodamajai (skat. att. 13). Tas liecina par to, ka izvēlētie praimeri ir bijuši pareizi, ka sekvenēšanas reakcija ir bijusi precīza.
Sekvenētajiem paraugiem vairākās vietās ir redzams tas, ka abu sekvence nesakrīt ar datu bāzes sekvenci (atzīmētās vietas, 13 att.). Tas varētu būt saistīts ar to, ka ir ieviesušās kādas punktveida mutācijas, vai nu PCR mutaģenēzes rezultātā vai arī cDNS klona sekvence pilnībā neatbilst datubāzei. Sakarā ar šīm neatbilstībām bakalaura darba ietvaros ir nepieciešams pārbaudīt izejas plazmīdas pXL 3-4 sekvenci, jo no E.Truves laboratorijas saņemtais klons nav ticis pilnībā sekvenēts. Sekvenēšanas rezultāti tika analizēti ar datorprogrammu - ContigExpress, kā arī Internetā atrodamās datu apstrādes programmas.
3. Potenciālie CfMV cDNS vektori
Konstruējot vektoru pJIT-CfMV-1 (skat. att. 14), vektors pJIT-60 vispirms tika griezts pa BamHI saitu, kam aizpildīja 5 galu ar Klenow fragmentu un pēc tam šķēla ar SalI. Iegūto fragmentu ekstraģēja no gēla pēc MBI, Fermentas DNS ekstraģēšanas kita. Plazmīda pXL 3-4K tika šķelta ar SalI un KspAI, iegūts ~ 4,0 kb garš fragments, ko ekstraģēja no gēla. Abus fragmentus saligēja un transformēja DH5a kompetentajās šūnās. Tika iegūti 11 miniprepi, kuru lizātus pārbaudīja vai tajos nav plazmīdas.
Sekvence:
Reversā ķēde BamHI c AGAgTGGgGTGGGGAGTCTGTCATGGCTGCCGCCATTGCGACGGATATGAGGAGAGCTAGC
No datu bāzes GGATCCCCATACATATCAGAGTGGG.TGGGGAGTCTGTCATGGCTGCCGCCATTGCGACGGATATGAGGAGAGCTAGC
Tiešā ķēde GGATCCCCATACATATCAGAGTGGG.TGGGGAGTCTGTCATGGCTGCCGCCATTGCGaCGGATATGAGGAGAGCTAGC
Reversā ķēde TTTGACCAGCTCATCCTGCTCATCAACATCACGGACTTGCAGTTTCCAACAACAGAACAACCCATTCTTGGTCACCCT
No datu bāzes TTTGACCAGCTCATCCTGCTCATCAACATCACGGACTTGCAGTTTCCAACAACAGAACAACCCATTCTTGGTCACCCT
Tiešā ķēde TTTGACCAGCTCATCCTGCTCATCAACATCACGGACTTGCAGTTTCCAACAACAGAACAACCCATTCTTGGTCACCCT
Reversā ķēde CAACCAACTAACCTCGGTGCTATTCTGTCCTGCCACGGCTGTAGCAGCAATTCCACGTTACACTGTAAGAGAAATCGC
No datu bāzes CAACCAACTAACCTCTGTGCTACTCTGTCCTGCCACGGCTGTAACAGCAATTCCACGTTACACTGTAAGAGAAATCGC
Tiešā ķēde CAACCAACTAACCTCGGTGCTATTcTGTCCTGCCtCGGCTGTAGCAGCAATTCCACGTTACACTGTAAGAGAAATCGC
Reversā ķēde TTGATACTTTGAAGCCTTTGGGCAGGGATGGAATCaCAACGGTCTCGCGGAATTCACAGATATCACAGTGAACTGTCA
No datu bāzes TAGATACTTTGAAGCCTTTGGGCAGGGATGGAATCACAACAGTTTCGCGGAATTCACAGATATCACAGTGAACTGTCA
Tiešā ķēde TTGATACTTTGAAGCCTTTGGGCAGGGATGGAATCACAACGGTCTCGCGGAATTCaCAGATATCaCAGTGAACTGTCA
Reversā ķēde AGCTCACAGGACCTCTGATCTTCAACCAGCGATGTTTGTTGGACTCACACCGGAACAGAACAACTTCAACGCCTTGGC
No datu bāzes AGCTCACAGGACCTCTGATCTTCGACCAGCGATGTTTGTTGGACTCACACCGGAACAGAACAACTTCAACGCCTTGGC
Tiešā ķēde AGCTCaCAGGaCCTcTgAtcTTCAACCAGCGATGTTTGTTGGACTCACaCCGGAACAGAACAACTTCAACGCCTTGGC
Reversā ķēde GGAAGAAGCACGAGCGGACGGGAATGGACTTGACAATTGTGGAATCGCCTGTCAACAAATCGTCGCTCGTGTAGCACT
No datu bāzes GGAAGAAGCACGAGCGGACGGGAATGGACTTGACAATTGTGGAATCGCCTGTCAACAAATCGTCGCTCGTGTAGCACT
Tiešā ķēde GGAAgAAGCACGAGCGGACGGGAATGGACTTGACAATTGTGGAATCGCCTGTCAACAAATCGTCGCTCGTGTAGCACT
Reversā ķēde TGAACGGTTATAAAACCAGGGGGAGGTTCGCACATCTAAGCTACTTCTCAACCTCTGATTCGGGAGGGGAACGATAAC
No datu bāzes .GAACGGTTATAAAACCAGGGGGAGGTTCGCACATCTAAGCTACTTCTCAACCTCTGATTCGGGAGGGGAACGATAAC
Tiešā ķēde .GAACGGTTATAAAACCAGGGGGAGGTTTGCAC..ATAAGCTACTTCTCAACCTCTGATTCGGGaGGGGaACGaTAAC
KspAI
Reversā ķēde AGTGTGTCTTTCTTCGCACTATTATCGTTAACGGCGCC
No datu bāzes AGTGTGTCTTTCTTCGCACTATTATCGTTAACGGCGCC
Tiešā ķēde aTTGCGTCTTTcttCgCAC...tATGgTTAAg..Ggcc
13. attēls. Sekvenču salīdzināšana. Iezīmētie rajoni vietas, kur nesakrīt tiešās un reversās DNS ķēdes sekvence ar datu bāzē esošo sekvenci; izceltie rajoni restrikcijas saiti, kuri tika gan izmantoti klonēšnā, gan plazmīdu atlasē (saits KspAI ar PCR ievadītais saits); mazie burti sekvences minorā zona.
Figure 13. Sequences comparison. Marked area places where discord direct and reverse DNA chain sequences with data base sequence; Display area restriction sites which were used both in cloning and in plasmide selection (site KspAI with PCR entered site); small caps sequins minor zones.
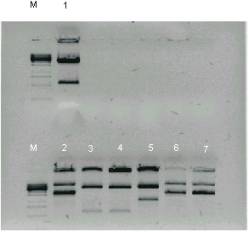
Analizējot uz 0,8 % agarozes gēla, varēja redzēt, ka ne visas plazmīdas ir liela izmēra (skat. att. 15). Garākās plazmīdas tika pārbaudītas ar restritāzi PvuII. No paraugiem mūs interesējošais bija tikai viens paraugs Nr. 11. Tam tika novērota pareizas restrikcijas vietas trīs fragmenti 1,7 kb, 1,8 kb, 4,3 kb gari (skat. att. 16).
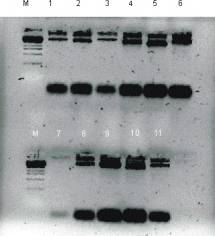
15. attēls. Miniprepu apskate 0,8 % agarozes gēlā 16. attēls. Paraugu pārbaude ar restriktāzi
uz plazmīdu pJIT-CfMV-1 (M marķieris, 1, 2, 3, 4, PvuII, lai pārliecinātos vai tā ir plazmīda
5, 6, 7, 8, 9, 10, 11 paraugi) pJIT-CfMV-1 (M marķieris, 1, 2, 3, 11 -
Figure 15. Miniprep examination in 0,8 % agar gel for paraugi, 11 pareizais klons)
plasmide pJIT-CfMV-1 (M marker, 1, 2, 3, 4, 5, 6, Figure 16. Samples examination with
7, 8, 9, 10, 11 samples) restriktasePvuII for assure that it is
plasmide pJIT-CfMV-1 (M marker, 1, 2,
3, 11 samples, 11 right clone)
Šai konstrukcijai tika novērota strukturālā nestabilitāte. Pēc atkārtotas transformācijas un miniprepu pagatavošanas, un plazmīdu analizēšanas 0,8 % agarozes gēlā, tika konstatēts, ka to garums ir mazāks. Griežot ar restriktāzēm, konstatējām, ka dod tikai vienu fragmentu. Tika noskaidrots, ka šai plazmīdai ir tieksme uz delēcijām, jo konstatējām, ka ir deletēts praktiski viss ieklonētais cDNS fragments. Šo plazmīdas nestabilitāti varētu izraisīt iespējamās rekombinācijas promotora, terminatora un cDNS starpā.
Tā kā konstrukcija - pJIT-CfMV-1 izrādījās nestabila, tika izvēlēta cita klonēšanas stratēģija.
Par vektoru tika ņemta plazmīda pXL 3-4K, kas tika šķelta pa PvuI un KspAI saitiem. Taču vektoram pXL 3-4K tika atrasts vēl viens PvuI saits. Tāpēc vektors tika šķeltas ar daļējo restrikcijas metodi, un no 0,6 % agarozes gēla tika ekstraģēts garākais fragments ~ 5,7 kb. Inserta fragments ar diviem 35S promotoriem tika ņemts no plazmīdas pJIT-60. Vispirms pēc šķelšanas ar BamHI, fragmenta 5 gals tika aizpildīts ar Klenov fragmentu un fragments tika iegūts izgriežot ar PvuI (PvuI saits abām plazmīdām atrodas ampicelīna rezisteces gēnā). pJIT-60 izolētā fragmenta garums ~ 1,3 kb.
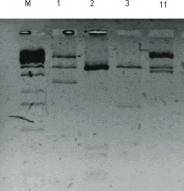
Abi fragmenti tika saligēti un iegūtā konstrukcija p34K-2x35S (skat. att. 17) tika transformēta XL1 šūnās. Tika iegūtas 7 kolonijas, no kurām iegūtie miniprepi tika pārbaudīti vai tie nesatur plazmīdas. Tika konstatēts, ka 6 miniprepi satur plazmīdas (skat. att. 18). Pirmais un otrais minipreps saturēja maza izmēra plazmīdu, bet trešais, ceturtais, piektais un sestais minipreps saturēja gara izmēra plazmīdas. Tās tika pārbaudītas ar restriktāzēm SspI,BamHI, EcoRI, PvuII (skat. att. 19).
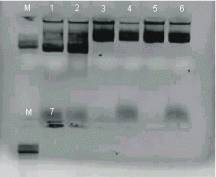
18. attēls. Miniprepu apskate 0,8 % agarozes 19. attēls. Paraugu pārbaude ar restriktāzēm SspI,
gēlā plazmīdai p34-2x35S (M marķieris, 1, BamHI/EcoRI un PvuII
2, 3, 4, 5, 6, 7 paraugi) (M marķieris, 3, 4, 5, 6 paraugi)
Figure 18. Miniprep examination in 0,8 % agars Figure 19. examination of samples with restriktase
gel for plasmide p34-2x35S (M marker, 1, 2, SspI, BamHI/EcoRI and PvuII
3, 4, 5, 6, 7 samples) (M marker, 3, 4, 5, 6 samples)
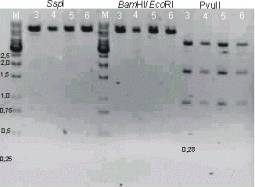
Tā kā SalI saita klātbūtne cDNS vektorā ir nepieciešama vektora sagatavošanai augu inficēšanai, papildus pārbaudē tika izmantota šī restriktāze. Visi četri kloni tika šķelti ar restriktāzēm SalI/EcoRI un SalI/Kpn2I (skat. att. 20).
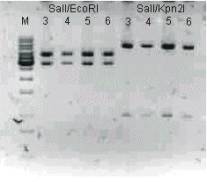
20. attēls. Paraugu pārbaude ar restriktāzēm SalI/EcoRI un SalI/Kpn2I, lai pārloecinātos par SalI saita esamību plazmīdā (M marķieris, 3, 4, 5, 6 paraugi, visi satur SalI saitu)
Figure 20. Examination of samples with restriktases SalI/EcoRI and SalI/Kpn2I for SalI sit dedication in plasmide (M- marker, 3, 4, 5, 6 samples, all contain sit SalI)
Restrikcijas analīzes rezultāti parādīja to, ka visi četri kloni satur pareiza garuma un skaita restrikcijas fragmentus.
Iegūtā konstrukcija p34K-2x35S ir CfMV infekciozās cDNS vektora kandidāts, kas tiks izmantots augu inficēšanai un rezultāti tiks iekļauti bakalaura darbā. Šī konstrukcija ir stabilāka par pJIT-CfMV-1, jo pēc atkārtotas E.coli šūnu kultivēšanas netika novērota plazmīdas nestabilitāte. Augu inficēšanai konstrukcija tiks šķelta pa SalI saitu un mehāniski ievadīta augu auzu - lapās pēc Scholthof, 1999 metodes.